
トップページ (2電子原子も含む正確な新ボーア模型)
タンパク質相互作用プログラムマニュアル。
「万物の理論」の失敗。
(Fig.1) 何が 疎水性 (親水性) を決定するのか?

基本的に ロイシンやフェニルアラニンなどの疎水性アミノ酸は タンパク質内部に存在する。
一方 グルタミン酸やアスパラギンなどの親水性アミノ酸は タンパク質表面に存在して 水分子と接している。
その主要な理由の1つが 水素結合だと 彼らは主張している。
しかし 例えば なぜ " C-H ---- O " の組み合わせは 水素結合になれないのだろうか?
( " N-H ---- O " や " O-H ---- O " は 水素結合の一般的な形であるにも関わらず。 )
このページでは、タンパク質相互作用面を調べることにより、これらの本当の理由を見つけることにする。
(Fig.2) 2つのタンパク質間の状態は?

ここでは タンパク質構造を調べるのに 次のサンプル JAVA プログラムを使う。
サンプル JAVA プログラム ( PDB タンパク質 - 4 量体 ).
最初に このマニュアルを読んで欲しい。
まず 次の小さなタンパク質2量体からスタートする。
このタンパク質は 光感受性で 162 アミノ酸 × 2 の 小さなタンパク質2量体である。
そのため このタンパク質からスタートするのが適切と思われる。
(Fig.3) コマンドプロンプト画面。

プログラム実行後、特に短い原子間距離のみ画面上に表示される。
O-C、 O-N、 O-O、 N-C、 C-C、 N-N などの原子間距離が 2.6 Å 未満のとき ( mixed subunit では 3.0 Å 以下 )、この原子位置をプログラム原子番号で示す。
例えば、 THR-N.39=THR-O.42>2.4536 は トレオニンの窒素 (= 原子番号, 39 ) と トレオニンの酸素 (= 42 ) 間の距離が 2.4536 Å であることを示している。
このマニュアルに示したように、各原子近くの固定領域は除いている。
"mixed subunitAB" の項では、例えば SER-O.242=TYR-O'.1500>2.4448 は サブユニットA のセリンの酸素と サブユニットB のチロシンの酸素間の距離が 2.4448 Å であることを示している。
点 ' は、この原子が アミノ酸側鎖にあることを表している。
お気づきだと思われるが、基本的に 原子間距離を 2.6 Å 未満に指定すると、
O-O ( もしくは O-N ) 結合しか見ることができない。
どうして C-C、 C-N、C-O、 N-N などは この距離以内では見られないのだろうか?
(Fig.4) A と B サブユニットの断面図。

このマニュアルに示したように、このプログラムは
"x-width"、 "y-width"、 "z-width" ボタンを使用することによって 任意の面の断面を表示することができる。
( 任意の方向の断面の width (幅) を自由に変更できる。 )
Fig.4 では、x-z (左), y-z (右) 平面で、 "y-width = ±5.0 Å" の条件が選択されている ( zoom=50 で )。
また 任意の2つの粒子を続けてクリックすると、その間の距離が テキストボックス内に表示される。
基本的に フェニルアラニン、アラニン、ロイシン、などの疎水性のアミノ酸は 互いに接している ( チロシンの疎水部分も含めて )。
また チロシンの先端の酸素原子は 水素結合によって B サブユニットのアスパラギン酸の酸素に近づいている。
A と B サブユニットの他の部分は ある一定の距離を保ったまま 互いに相補的に きれいに並んでいるのが分かる。
このタンパク質は転写に関係している ニッケル活性の 2量体タンパク質である ( 152 アミノ酸 × 2 ).
(Fig.5) コマンドプロンプト画面。

プログラム実行後、短い原子間距離の場所が表示される。
O-C, O-N, O-O, N-C, C-C, N-N の原子間距離が 2.6 Å 未満 ( mixed subunit では 3.0 Å 未満 ) のとき、この位置がプログラム原子番号によって示される。
お気づきのとおり、このケースでも 2.6 Å 未満の距離を指定したとき、
O-O ( もしくは O-N ) 結合のみが見られる。
( このページで述べたように、反発力により 約 2.5 Å が O-O や O-N の非共有結合の 下限極限と考えられる。 )
再び、 C-C、 C-N、 C-O、 N-N は この結合距離に見られない。
(Fig.6) A と B サブユニットの断面図。
このマニュアルに示したとおり、 "x-width" ボタンを用いて このタンパク質の断面を見る。
Fig.6 では、x-z (左), y-z (右) 画面で "x-width = ±5.0 Å" の条件が選択されている。
上記の2量体のように、原子間 3.0 Å 未満を指定したとき、 基本的に 酸素-酸素 (もしくは 窒素 ) 結合のみしか見られない。
A-B サブユニット接触面の C-C 原子間距離は 約 4.0 Åで、これはつまり H-H 長が 約 2.0 Å であることを意味している。
このページで述べたように、C-O (= H2O の酸素 ) 間の反発力は C-C 間よりも強くなる。
この C-C 距離は タンパク質内で H2O 分子を排除する 上限極限付近の距離と考えられる。
ニッケル (= Ni ) などの金属は その長いドブロイ波のために 強い正電荷の性質を示す (= 原子核が 露出しているため )。
そのため 窒素や酸素などの 結合に使われていない余剰の電子をもつ原子のみが ニッケルに近づくことができる。
このタンパク質は加水分解に関係している 484 アミノ酸 × 2 の 長い2量体タンパク質と ナトリウムイオンからなる。
そのため スタートアップに少し時間がかかる。
(Fig.7) コマンドプロンプト画面。

このケースでも 2.6 Å 未満の距離を指定したとき、
O-O ( もしくは O-N ) 結合のみが見られる。
また A と B サブユニット間の距離を 3.0 Å 以内としたとき、C-C、 C-N、 C-O、 N-N は この結合距離にほとんど見られない。
(Fig.8) A と B サブユニットの断面図。

このマニュアルにあるとおり、 "z-width" ボタン ( zoom = 50 ) を用いて このタンパク質を切断する。
Fig.8 では、 x-y (左)、y-z (右) 画面で、 "z-width = ±5.0 Å" の条件を選択している。
上記の2量体のように、基本的に 3.0 Å以内の距離のとき、酸素-酸素 ( もしくは 窒素 ) 間の結合しか見られない。
疎水性タンパク質の炭素原子は H2O 分子の酸素の反発力を避けて 他の疎水性タンパク質と2量体を形成しようとする。
(Fig.9) A と B サブユニットの断面図。

ナトリウムイオンが このタンパク質に含まれている。
上記の金属同様、グルタミン酸、アスパラギン酸、ヒスチヂン等の 酸素、 窒素のみ 正のナトリウムイオンに引きつけられる。
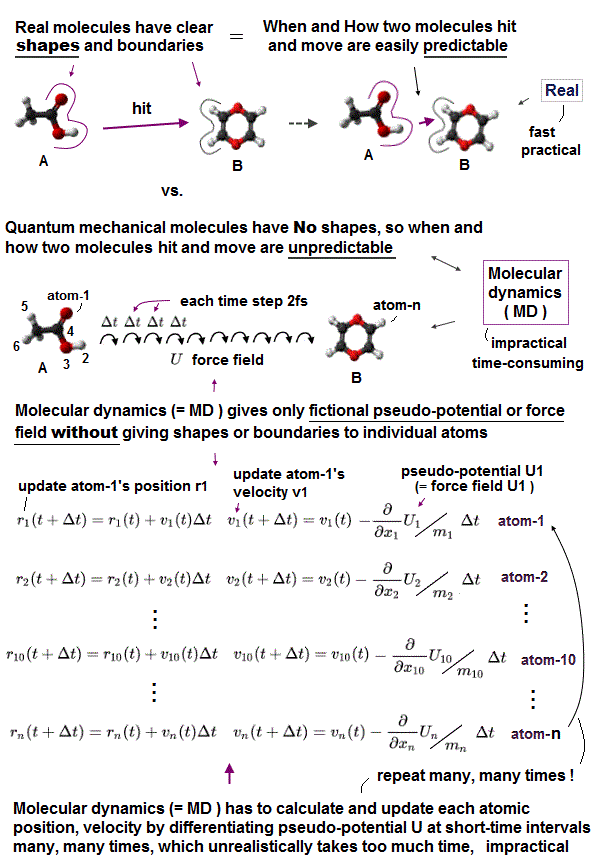
(Fig.10) H -- O-H 水素結合。
このページでは、水素原子の反発力が非常に 弱く、この水素の性質が水素結合に関係していることを示した。
H -- O-H 水素結合長は 約 2.60 - 2.70 Å である。
このプログラム実行後、 "O" と "H" を A、 B 原子として選び、その原子間距離を "1.60 Å" (= 16000 MM ) にする。
そして Fig.10 のように "O" 原子を x-y 平面で 45°回転させる ( "45" を入力して "x-y ang" ボタンをクリックする )。
このケースでは、結合エネルギーは 正 (= 0.004 eV ) になり、 O-H 結合は 安定になる。
( このプログラムでは 結合エネルギーが "正" のとき、この結合が安定になる。)
また 原子間反発力は ほぼゼロである ( FX = -76, FX = 16 )。
( これらは ほぼゼロだが わずかに "反発"しているため、 O-H や N-H 結合長は これより短くなれない。 )
| N - A 長 | 1.90 Å | 1.80 Å | 1.70 Å | 1.60 Å | 1.50 Å | 1.40 Å | 1.30 Å |
|---|---|---|---|---|---|---|---|
| O - H | -0.011 | -0.009 | -0.004 | +0.004 | +0.022 | +0.054 | +0.113 |
| O - C | -0.205 | -0.190 | -0.158 | -0.095 | +0.020 | +0.232 | +0.612 |
| O - O | -0.261 | -0.266 | -0.265 | -0.252 | -0.215 | -0.122 | +0.092 |
Table 1 に示した通り、O-H 結合のみが この距離 (= 1.60 Å) のときに安定である。
なぜなら 水素原子核が 小さく かつ 露出しているからである。
C や O などの他の原子では、価電子や原子核間の反発力が優性になる。
すべての結合において、結合長が短くなるにつれて、その結合は共有結合様になって 安定になる。
(Fig.11) O-O、O-C における反発力
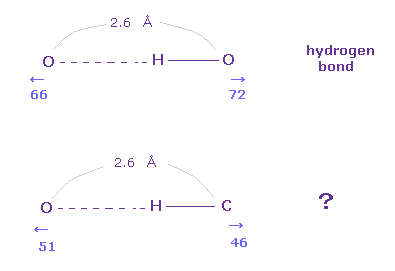
上のセクションで述べたとおり、原子間距離 2.6 Å あたりの O-C 結合は 様々なタンパク質においても ほとんど見られない。
Fig.11 に示したように、このプログラムを用いれば O-O の反発力が O-C よりも 強くなることが分かる。
この事実にも関わらず、どうして O-C 長 (= 非共有結合 ) は 2.6 Å ほどに短くなれないのだろうか?
これらの長い距離では、正の酸素原子核の反発力が優勢になる。
上のタンパク質では A と B サブユニット間の O-C 長は 通常 3.6 Å 以上 である。
(Fig.12) 炭素のすべての価電子 (= 結合状態 ) は動けない。

基本的に ある正電荷が近づいたとき、価電子のいくつかは この正電荷の方へ動いて この反発力をキャンセルしようとする。
しかし炭素の場合では、4つの価電子のすべては すでに 他の原子核に結合していて 動くことができない。
( 正電荷が強すぎて 共有結合を切断させるなどのケースは除いて。)
炭素の価電子の1つが 酸素の方へ動いたとしたら、それは 他の原子核が反対の方向へ 動くことを意味している。
しかし 共有結合は 非常に強いため、この現象は 起こりがたい。
(Fig.13) 酸素のフリーの価電子が 他の正の電荷方向へ移動する。

一方、酸素は6つの価電子を持ち、そのうち 4つは非結合の フリーの状態である。
これらの自由な電子は 常に 他の正電荷の原子核を探して そちら方向へ動こうとする。
この動きが 原子核間の反発力を キャンセルする。
(Fig.14) 酸素の電子の "up" と "down" の電子の引力。

基本的に 酸素の up と down の価電子の引きつけ力は非常に強い。
Fig.14 に示したように 電子と酸素原子核間の力が "F" のとき、 それに垂直な 引きつけ力は F sinθ になる。
θ に対する その微分は cosθ になる。
Fig.14 に示したように この微分 (= 力の増加率 ) は この θ が ゼロ近くのときに最も大きくなる。
他の原子核の反発力が強くなりすぎると、この θ は 90°に近くなる。
そのため この時は up と down の電子間反発力の増加のほうが 引きつけ力の増加よりも大きくなってしまう。
(Fig.15) フリーの "up" と "down" の N の電子が 他の原子核方向へ移動。

窒素 (= N ) 原子においても、 "up" と "down" の電子がフリーで 他の正電荷を探している状態である。
結果、 O-O や O-N 長は 2.5 Å と短くなることができ、 O-C 長は なれないことになる。
( もちろん C 近くで 強固な共有結合で繋がっている状態を除く。 )
このページで述べたように、酸素が この長さよりも水素に近づくとき、O と H 間の反発力は強くなり、これをブロックする。
(Fig.16) ミオグロビンの鉄付近の原子。

この PDB ファイル ( 1MBO ) (= oxymyoglobin ) を用いて、 鉄 (= FE ) 付近の原子は 必ず 窒素もしくは酸素になることがわかる。
Fig.16 では、 "PDBat" の条件で FE 原子 "1276" を選択し、"center" ボタンをクリックする (= 7 Å の距離で )。それから y-width を ± 4.0 Å にする。
このプログラムを持ちれば、 原子間距離が 約 1.9 Å あたりのとき、FE (= 鉄 ) の 窒素に対する反発力は 約 1000 ほどであることが分かる。
( FE 原子の価電子の1つを窒素方向へ向けるため -45°回転させた後。 )
この反発力は 窒素の "up" と "down" の電子を 少し 鉄方向へ移動させて キャンセルさせることができる。
もちろん、炭素原子には このようなことができない。
水素原子は この FE-O の間に入れない。なぜなら 鉄の強い反発力が 水素原子核を反発し、水素の電子を引きつけつため N-H 結合を不安定にさせてしまうからである。
(Fig.17) 炭素は 酸素を避けて 他の C-H のほうへ近づく。

このプログラムを用いれば、 C-C 間の反発力が C-O 間よりも弱いことが分かる。
そのため 炭素原子 (= C ) は 水の酸素原子を避けるようにして 他の C-H 残基方向へ近づいていく。
Fig.12 (= C の価電子 ) と Fig.17 が 疎水性を示す 本当のメカニズムである。
サブユニット A と B のタンパク質間の C-C の長さは 約 4.0 Å である。
そのため C と C 間の小さな反発力のため、それらは この長さよりも互いに近づくことができないと言える。
この C-C 長では H2O 分子が その間に入っていくスペースがほとんどない。
( 水素原子も考慮して。 )
これはつまり 2つのタンパク質の疎水性部分は 接触面で いわゆる陰圧によって 互いにくっついていると言える。
(Fig.18) セルロース内の水素結合が H2O 分子をブロックする。

セルロースは 多くの O-H 残基を含んでいるため 完全に親水性分子と言える。
しかし セルロースは水に不溶性である。
なぜなら セルロース内の H と OH 残基が 互いに水素結合を形成するからである。
そのため ( 2つの ) H2O 分子が その間に入っていくスペースがない。
これが 親水性のセルロースが 水に不溶性な理由である。
(Fig.19) 112 (= G ) と 113 (= C ) の残基。

この PDB ファイルは TATA-結合 タンパク質-DNA 複合体である。
このページに示したように、 DNA ( もしくは RNA ) 部分が PDB ファイルの最初の位置にあるとき、この DNA 部分が サブユニット B になる。
このプログラムでは サブユニット B を指定するとき、
"10000" を原子番号などにつける。
( Fig,19 では、 10112 から 10113 の残基を表示している。 )
(Fig.20)

上のケースのように、基本的に O-N もしくは O-O 長は 水素結合のために 容易に 2.5 Å になる。
このケースでは、mixed subunit は タンパク質と DNA 複合体である。
これらの相互作用においても、N-O 長は 2.5 Å と短くなれる。
PDB ファイル (2J0X), E.coli Aspartokinase
このタンパク質は 450 アミノ酸 × 2 の 大きなタンパク質である。
このタンパク質においても、 O-O もしくは O-N は 容易に 2.5 Å になれる。
このタンパク質は 318 アミノ酸 × 2 のタンパク質である。
このタンパク質においても、 O-O もしくは O-N は 容易に 2.5 Å になれる。
( 同じアミノ酸内で 短い O-C 長が見られることもある。 )

2013/8/8 updated This site is link free.